 |
 |

Non validation de facteurs génétiques de risque rapportés dans le syndrome coronaire aigu dans une étude de réplication à large échelle
Thomas M. Morgan, MD;
Harlan M. Krumholz, MD, MS;
Richard P. Lifton, MD, PhD;
John A. Spertus, MD, MPH
RÉSUMÉ
 |  |
Contexte Compte tenu des nombreux, mais non totalement pertinents,
rapports sur les variants génétiques associés aux
syndromes coronaires aigus (SCA), il est nécessaire de valider de
façon approfondie les génotypes de susceptibilité dans
les syndromes coronaires.
Objectif Réaliser une validation approfondie des facteurs
génétiques potentiels de risque associés aux SCA.
Schéma, environnement et participants Dans une recherche
systématique de la littérature sur les articles publiés
avant le 10 mars 2005, nous avons identifié des variants
génétiques précédemment rapportés comme
étant des facteurs significatifs de susceptibilité au
développement d'une athérosclérose ou d'un SCA. En
restreignant nos analyses à des patients blancs pour diminuer la
possibilité de confusion due au mélange de races, nous avons
identifié 811 patients qui s'étaient présenté
entre mars 2001 et juin 2003 avec un SCA dans les deux hôpitaux
universitaires de Kansas City, Missouri. Durant 2005-2006, nous avons
génotypé 811 patients avec 650 témoins appariés
pour l'âge et le sexe pour 85 variants de 70 gènes et avons
essayé de répliquer les associations précédemment
rapportées. Nous avons exploré d'autres associations possibles
sans hypothèse antérieure de modèle spécifique de
risque et utilisé le test Sign pour chercher les faibles
associations.
Principaux critères de jugement Comparer un variant
pré-spécifié de gène associé à un
risque de SCA chez les cas et les témoins. Un excès
d'associations impliquant que certains pourraient être associés
aux SCA.
Résultats Parmi les 85 variants testés, un seul
génotype putatif de risque (-455 variant du promoteur du
β-fibrinogène) était statistiquement significatif (P=0.03).
Seuls 4 autres gènes étaient positifs dans l'analyse du
modèle libre. Aucune association n'a dépassé la
fréquence liée au hasard, compte tenu du nombre de comparaisons.
Finalement, seuls 41 des variants pré-spécifiés de risque
sur 84 étaient plus fréquents chez les cas que chez les
témoins (avec une égalité), ce qui représente un
taux de gain de 48.8% (intervalle de confiance à 95%, 38.1%-59.5%) pour
les génotypes de risque collectif (P=0.91, test Sign).
Conclusions Nos résultats nuls ne sont pas en faveur de
l'hypothèse qu'un des 85 variants génétiques
testés est un facteur de risque de susceptibilité de SCA. Ces
résultats mettent l'accent sur la nécessité d'une
réplication solide des facteurs génétiques putatifs de
risque avant leur utilisation en clinique.
JAMA.
2007;297:1551-1561
Des preuves incontestables provenant d'études chez les
jumeaux et des études épidémiologiques suggèrent
une base génétique de l'athérosclérose et des
syndromes coronaires aigus (SCA), dont l'angor, les infarctus du myocarde sans
sus-décalage du segment ST (NSTEMI) et avec sus-décalage du
segment ST
(STEMI).1,2
A ce jour, principalement dans des études castémoins, de
nombreux gènes candidats ont été impliqués comme
facteurs potentiels de risque cardio-vasculaire, mais peu, s'il y en a, ont
été reconnus
définitivement.3-5
Les facteurs affaiblissant la validité des rapports
précédents incluent des tailles d'échantillon
inappropriées, des comparaisons multiples de sous-groupes et des biais
de
publication.4
Avant d'être d'utilité clinique, les facteurs
génétiques potentiels de risque devraient être de
façon idéale reproductibles en masse dans de larges populations
bien définies de
patients.6
Mais à ce jour, aucune validation approfondie des variants
génétiques potentiellement associés aux SCA ou à
l'athérosclérose n'a été rapportée. En
conséquence, nous avons premièrement cherché à
identifier les associations génétiques aux SCA en recherchant
systématiquement dans la littérature les variants
rapportés comme étant associés à un IDM, à
l'angor instable ou l'athérosclérose. Nous avons alors
essayé de valider ces risques génétiques putatifs au sein
d'une large étude cas-témoin.
METHODES
Gènes candidats
Nous avons fait une recherche PubMed, dans les bibliographies d'articles
originaux et de revues, des manuscrits publiés avant le 10 mars 2005
qui avaient rapporté des associations statistiquement significatives
entre des génotypes spécifiques et
l'athérosclérose coronaire ou les SCA (une liste des articles
est disponible sur demande auprès des auteurs). Les mots-clés de
la recherche MEDLINE incluaient: gene, genetic, polymorphism, myocardial
infarction, atherosclerosis, coronary heart disease, et coronary artery
disease. Les rapports étaient inclus s'ils indiquaient une association
positive significative, avec une valeur de p rapportée par
l'investigateur de 0.05. Un total de 96 variants génétiques
polymorphiques de 75 gènes a été mis en évidence
et inclus (TABLEAU 1 et
TABLEAU 2). Onze de ceux-ci ont
été exclus en raison de leur échec au test de
génotypage multiplex.
|
|

|
|
Tableau 1.. Validation des comparaisons des génotypes prédéfinis
du risque chez les cas vs témoins
|
|
|
|
|

|
|
Tableau 2.. Fréquences de génotype et valeurs de p chez les cas ayant un
syndrome coronaire aigu et les témoins
|
|
|
Description des cas et des témoins
Huit cent onze patients blancs ayant des ancêtres européens
avec des antécédents de SCA ont été
identifiés lors de leur venue dans deux hôpitaux de Kansas City,
Missouri, (Mid-America Heart Institute et Truman Medical Center), entre mars
2001 et juin 2003. Les définitions de référence des SCA
ont été utilisées pour le diagnostic des patients ayant
soit un IDM soit un angor
instable.92,93
Un infarctus du myocarde était défini par un test positif
à la troponine réalisé dans le contexte de
symptômes et de modifications de l'électrocardiogramme (à
la fois modifications de type sus-décalage du segment ST et sans
décalage du segment ST) compatibles avec un IDM. Les diagnostics
d'angor instable étaient confirmés par la conjonction de l'avis
de 3 médecins revoyant le dossier, si les patients avaient des tests
négatifs pour la troponine et l'un des éléments suivants:
angor nouvellement installé (<2 mois) au moins de classe III de la
classification de la Canadian Cardiovascular Society, un angor de repos
prolongé (>20 minutes), une aggravation récente (<2 mois)
de l'angor ou un angor qui était apparu dans les deux semaines
après un
IDM.93 Parmi
les patients troponine négative ayant un angor instable, 203 (92.7%)
ont eu une cathétérisation cardiaque, un test d'effort avec
scintigraphie ou un échocardiogramme de stress pour corroborer le
diagnostic.
Chaque patient hospitalisé participant ayant un SCA était
interrogé pour déterminer les variables, comme le tabagisme, la
consommation d'alcool, les antécédents familiaux (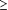 1 parent
de premier degré ayant eu un IDM ou une maladie coronarienne), et pour
avoir son consentement afin de réaliser un prélèvement
sanguin destiné à l'analyse génétique. Par
ailleurs, des résumés détaillés des dossiers
étaient réalisés pour recueillir les données
pertinentes biologiques et cliniques. 1 parent
de premier degré ayant eu un IDM ou une maladie coronarienne), et pour
avoir son consentement afin de réaliser un prélèvement
sanguin destiné à l'analyse génétique. Par
ailleurs, des résumés détaillés des dossiers
étaient réalisés pour recueillir les données
pertinentes biologiques et cliniques.
Au total, 1045 patients SCA (parmi lesquels 811 patients blancs ont
été inclus dans l'étude actuelle) ont été
d'accord pour participer et pour donner un échantillon de sang
destiné à l'analyse génétique. Les patients
déclaraient spontanément leur race/ethnie en choisissant un des
éléments descriptifs suivants fournis par les investigateurs:
blanc, blanc latino-américain, afroaméricain et
afro-américain non latinoaméricain. Les sujets témoins
appariés pour l'âge et le sexe étaient recrutés
à la consultation ambulatoire des malades externes de l'un des centres,
Saint Luke's Hospital à Kansas City. Ces patients avaient alors des
analyses systématiques et il leur était demandé de
répondre à un questionnaire définissant les facteurs de
risque cardiovasculaires et les comorbidités médicales. Les
témoins rapportant un SCA antérieur, un pontage aorto-coronarien
antérieur ou une intervention coronaire antérieure
percutanée étaient exclus. Pour minimiser l'impact potentiel
d'un mélange génétique, 650 témoins blancs
d'ascendance européenne qui n'avaient rapporté aucun
antécédent de maladie coronarienne ont été choisis
parmi les 1054 témoins potentiels. Les données des facteurs de
risque manquaient pour 9 témoins sains appariés sur le sexe et
l'âge, et 56 autres témoins appariés ont été
utilisés pour un haplotypage ALOX5AP.
Le protocole de recherche a été approuvé par les
comités de recherche institutionnels de chaque centre; tous les
participants de l'étude avaient fourni un consentement
éclairé écrit pour études cliniques et
génétiques.
Génotypage
L'ADN génomique était isolé (Gentra PUREGENE,
Minneapolis, Minn) à partir des échantillons sanguins et soumis
à une amplification du génome complet par déplacement
multiple du brin (Molecular Staging Inc, New Haven, Conn), à l'aide
d'un amorçage aléatoire et d'une polymérase
Phi-29.94,95
Un génotypage était réalisé à l'aide d'un
système Sequenom MALDI-TOF (Matrix Assisted Laser Desorption-Ionization
Time-of-Flight), utilisant un logiciel Spectrodesign pour la conception du
test (Sequenom, San Diego, Calif), et des méthodes de test
précédemment
décrites.96,97
Les variants des gènes étaient exclus de l'analyse s'ils ne
pouvaient être génotypés à l'aide du système
Sequenom en raison d'un échec persistant du test, défini par
moins de 95% des génotypes scorables après 4 cycles de
réaction multiplex. Onze tests ont été en
définitive
exclus.* Pour la rare
délétion de paire de base MEF2A 21-base pair (bp), les cas et
les témoins étaient génotypés par une
réaction en chaîne à la polymérase afin de
générer des amplicons de 152-bp non délétion ou de
131-bp de délétion suivis par une électrophorèse
sur gels d'agarose à 3%. Les délétions mises en
évidence étaient confirmées par un
séquençage direct des séquences d'ADN. En raison de sa
rareté, MEF2A a été analysée
séparément et en conséquence seuls les 84 autres ont
été soumis à la série complète d'analyses
statistiques. Une version 2.1 PHASE a été utilisée pour
évaluer les fréquences d'haplotypes pour
ALOX5AP.101,102
Analyse statistique
Les distributions génotypiques chez les cas et les témoins
ont été évaluées à la recherche d'une
déviation significative (P<0.05) de l'équilibre de
Hardy-Weinberg. Le nombre de départs a été
évalué par une simulation de Monte Carlo et comparé au
nombre qui aurait été obtenu de façon aléatoire
(Resampling Stats Inc, College Park, Md).
Dans l'analyse principale, chaque variant génétique
était prédéfini sur la base des rapports
déjà publiés et les fréquences des variants
liés au risque étaient comparées chez les cas et les
témoins à l'aide d'une extension Monte Carlo avec
itération 100 000 du test du
chi-2 (SPSS
13.0 Exact Tests, SPSS Inc, Chicago, Ill). Le terme statistiquement
significatif était réservé à une valeur de p
inférieure à un seuil de significativité dans toute
l'étude avec une correction de Bonferroni 0.05/84=0.0006). La
correction de Bonferroni étant conservatrice lorsqu'on l'applique
à une étude de réplication, le nombre total de toutes les
associations positives à un niveau p<0.05 était aussi
comparé au nombre obtenu de façon aléatoire dans 100 000
simulations. Un surplus d'associations positives par rapport aux estimations
aléatoires impliquait que certaines étaient réellement
associées aux SCA.
Nous avons ensuite comparé les distributions génotypiques
globales pour chaque site chez les cas et les témoins par un test du
chi-2 Monte Carlo. La puissance pour confirmer des associations
génétiques individuelles était déterminée
par une méthode basée sur la probabilité logarithmique
(Quanto
1.0).103,104
Enfin, en tant que mesure pour augmenter la puissance, le pourcentage
observé de variants prédéfinis du risque observés
comme étant même marginalement plus fréquents chez les cas
que chez les témoins a été évalué par un
test Sign. En prenant une hypothèse nulle, chacun des variants de
risque a une probabilité égale d'être plus fréquent
chez les cas, ou chez les témoins. Pour évaluer la puissance du
test Sign à détecter un excès d'associations
génétiques positives, même faibles (50 des 84 associations
positives confèrent une valeur de p = 0.08 avec le test Sign), nous
avons simulé un ré-échantillonage des génotypes
des 650 témoins et des 811 cas dans les 84 comparaisons
génotypiques, trouvant un rapport de cotes minimal détectable
assurant un niveau critique de probabilité avec un taux gagnant de
63.3% pour chacun des 84 variants de risque d'avoir au moins 50 gagnants
(positifs) avec une confiance de 80%.
RESULTATS
Les caractéristiques cliniques des 811 cas et des 650 témoins
sont décrits dans le TABLEAU
3 et les distributions de leurs génotypes sont
montrées dans le Tableau
2. La population des cas SCA incluait 308 (38%) STEMI, 284 (35%)
NSTEMI, et 219 (27%) patients ayant un angor instable. Les cas et les
témoins avaient une distribution de l'âge, du sexe et de l'indice
de masse corporelle similaires. Un antécédent familial de
maladie coronarienne ou d'IDM chez les parents de premier degré
était multiplié par 2.7 fois chez les cas de sexe masculin que
chez les témoins de sexe masculin et par 2.0 chez les cas de sexe
féminin par rapport aux témoins de sexe féminin.
|
|

|
|
Tableau 3.. Caractéristiques des 1461 participants blancs
génotypés pour 85 variants
génétiques*
|
|
|
Les cas de sexe masculin et de sexe féminin avaient
significativement plus de probabilité d'être toujours fumeurs et
d'avoir un diabète de type 2 mais avait moins de probabilité de
consommer au moins une boisson alcoolisée par mois. Les taux
d'hypercholestérolémie et d'hypertension artérielle
étaient plus élevés chez les cas de sexe féminin
que chez les témoins, sans différence significative chez les
hommes. Des antécédents de revascularisation étaient
présents chez 35.6% des nouveaux cas de SCA mais pas chez les
témoins.
Un total de 85 variants chez 70 gènes ont été
génotypés chez les cas et les témoins. Le taux global de
génotypes chez ces variants a été de 98.5%
(extrêmes, 95.0%-99.8%). Deux pour cent de tous les échantillons
ont été génotypés en aveugle deux fois pour chaque
marqueur en tant que mesure de la reproductibilité du génotype.
Parmi les 2511 génotypes répétés, 5 étaient
discordants, démontrant une reproductibilité de 99.8%.
Les tests de l'équilibre de Hardy-Weinberg ont
révélé qu'un variant dépassait cet
équilibre à la fois chez les cas et chez les témoins,
à un niveau de p<0.05; 7 le dépassaient chez les cas
seulement et 4 le dépassait chez les témoins seulement
(Tableau 1 et
Tableau 2). Cette observation
ne dépasse pas ce que l'on observerait de façon aléatoire
(4 violations attendues de façon aléatoire dans chaque groupe;
voir la section Méthodes) et en conséquence aucun n'a
été exclu de l'analyse à ce stade.
Par rapport aux paramètres de puissance, la fréquence moyenne
effective (ou 1-fréquence si q>0.5) chez les témoins des
variants putatifs de risque étudiés était de 0.20 et 58
(68.2%) étaient communs (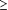 0.1), 25 (29.4%) étaient peu
communs (<0.1; >0.01), et 2 (2.4%) étaient rares ( 0.1), 25 (29.4%) étaient peu
communs (<0.1; >0.01), et 2 (2.4%) étaient rares ( 0.01).
Notre échantillon avaient une puissance de 80% pour confirmer, avec un
test chi-2
Monte Carlo, un risque relatif spécifique génotypique de 3.2
pour un variant rare (q=0.01), 1.4 pour un variant relativement peu commun
(q=0.1), et 1.25 pour un allèle commun (q=0.5). 0.01).
Notre échantillon avaient une puissance de 80% pour confirmer, avec un
test chi-2
Monte Carlo, un risque relatif spécifique génotypique de 3.2
pour un variant rare (q=0.01), 1.4 pour un variant relativement peu commun
(q=0.1), et 1.25 pour un allèle commun (q=0.5).
Nous avons testé si chaque variant putatif de risque montrait une
différence significative de fréquence entre les cas et les
témoins (Tableau 1). Un
rapport de cotes supérieur à 1 indique que le génotype du
risque se situait dans les fréquences élevées chez les
cas, et si tel était le cas, la différence de fréquence
du génotype était rapportée comme étant un nombre
décimal positif. Seul un variant génétique était
significatif au niveau de p<0.05, qui est le nombre le plus probable que
l'on obtiendrait de façon aléatoire seulement. Le
variant—455, qui se situe en amont du site d'initiation de la
transcription dans le gène ?-fibrinogène, répliquait
l'association originalement rapportée, avec un génotype GG plus
fréquent chez les cas que chez les témoins (fréquence,
66% chez les cas vs 61% chez les témoins; rapport de cotes, 1.27;
p=0.03). De plus, nous avons trouvé une délétion MEF2A
21-bp dans un cas et un témoin, confirmant que ceci était un
variant rare au sein de la
population.105
Plusieurs analyses supplémentaires ont été
réalisées. Lorsque les génotypes des cas et des
témoins ont été analysés par extension des tests
chi-2 2x3
à 100 000 simulations, 4 loci, RECQL2, THBS2, LIPC, et p22-PHOX,
étaient marginalement significatifs
(Tableau 2). Dans chaque cas,
le modèle du risque génétique spécifique donnant
une significativité était différent de celui
rapporté dans la littérature; en conséquence, ceci ne
peut pas être considéré comme des réplications
formelles et le nombre total d'associations positives n'est pas en
excès des estimations aléatoires.
Nous avons trouvé enfin que seulement 41 des 84 variants
pré-définis du risque étaient même marginalement
plus fréquents chez les cas que chez les témoins (à
l'exclusion d'une égalité, la rare délétion
MEF2A), représentant un taux gagnant de 48.8% (intervalle de confiance
à 95%, 38.1%-59.5%) pour les génotypes du risque collectif. Ce
pourcentage observé de gagnants n'est pas différent du
pourcentage attendu (50%) avec une hypothèse nulle (P=0.91). Le
Tableau 1 montre que les
différences absolues des fréquences du génotype de risque
entre les cas et les témoins (les signes négatifs signifiant que
le génotype putatif de risque était plus fréquent chez
les témoins que chez les cas) étaient faibles, avec une
différence médiane de -0.0003 et un maximum de 0.056 (β
fibrinogène).
COMMENTAIRES
 |  |
Nous n'avons pu confirmer en tant que facteurs de risque de SCA, 85
variants génétiques car aucun n'a été
validé sans équivoque dans cette large étude
cas-témoin chez 1461 participants. Dans l'analyse primaire, seul le
variant du promoteur -455 du β-fibrinogène) a été
nominalement statistiquement significatif (P=0.03). Parmi les 4 variants de
l'analyse secondaire qui répondaient aux seuils statistiques nominaux,
il y a eu un excès de variant différent que ce qui avait
précédemment rapporté chez les cas dans l'étude
originale, ce qui n'est pas en faveur d'une réplication. Nous pouvons
donc conclure que nos observations, dans ce large échantillon de
patients SCA et témoins bien définis, ne supportent pas
l'hypothèse que ce panel de variants de gènes contient en toute
bonne foi des facteurs de risque de SCA.
Nos observations arrivent à un point critique dans la
génétique des maladies complexes. Certains variants de
gène cardio-vasculaire (eg, ACE,AGT, AGTR1, ITGB3, F2, F5, MTHFR)
inclus dans notre étude peuvent déjà être
réalisés en clinique, pour des indications qui incluent de
façon claire un risque possible de SCA. Toutefois, nos observations
suggèrent que ces tests cliniques génétiques sont
prématurés et soulignent l'importance d'études robustes
de réplication sur des associations rapportées avant de les
appliquer aux soins cliniques. Ces non réplications incluent des
variants dans plusieurs études de profil. Par exemple, les haplotypes A
et B de la protéine activant la 5-lipoxygénase (ALOX5AP) ont
été rapportés dans une étude comme étant
associés aux IDM dans la population générale en Islande
et au
Royaume-Uni.17
Nous n'avons trouvé aucun haplotype associé à un SCA en
dépit des fréquences observées d'haplotype chez les cas
et les témoins ce qui est proche de ceux observés
précédemment dans la série totale des données au
Royaume-Uni (cas et témoins) (haplotype A, 0.165 vs 0.160; haplotype B,
0.062 vs 0.058). Bien que notre étude soulève des doutes
significatifs sur le panel collectif des facteurs génétiques
putatifs de risque, il n'infirme pas les études
précédentes. Les explications possibles de nos résultats
négatifs peuvent inclure: (1) résultats fauxnégatifs dans
notre étude; (2) associations faussement positives dans les
études antérieures; et (3) effets variés des variants de
risque dans des contextes génétiques différents.
Les résultats faux-négatifs comme explication
générale des observations nulles de notre étude sont
improbables compte tenu que la taille de notre échantillon est
substantiellement plus importante que l'ensemble des études
rapportées mis à part un petit nombre et que notre étude
avait une puissance permettant de déceler des risques relatifs
modestes. Sur la base d'un échantillon aléatoire (n=30)
d'articles inclus dans cette étude (1 par variant de gène), nous
avons estimé que le rapport de cotes moyen rapporté dans les
études positives était de 2.3 (extrêmes, 1.25-5.0),
indiquant que nous avions largement plus que 80% de puissance pour faire une
réplication de la plupart des rapports. Toutefois, des rapports
positifs isolés peuvent surestimer les risques
génétiques.5,6
Récemment, une méta-analyse de 14 gènes inclus dans notre
étude rapportait des rapports de cotes allant de 1.10 à 1.73
pour un risque
d'IDM.3 Il
est possible que des rapports de cotes très faibles soient attendus
dans des maladies génétiques complexes et que ni notre
étude ni la plupart des études précédentes n'aient
eu la puissance nécessaire. En conséquence, nous avons
augmenté notre puissance, en utilisant le test Sign, pour
détecter un surplus aussi faible que 16 facteurs
génétiques de risque faiblement positifs parmi la série
entière que nous avons génotypée (84-16=50, le nombre
requis pour un test Sign significatif), correspondant à un rapport de
cotes moyen ou plus élevé compte tenu de la taille de notre
échantillon et de la fréquence moyenne du génotype du
risque. L'absence d'effet génétique seulement dans notre cohorte
est improbable. Les cas montraient une multiplication par deux
d'antécédents familiaux de SCA, compatibles avec un effet
génétique contribuant aux phénotypes de cette cohorte. De
plus, une homozygotie codant pour un résidu d'arginine en position 158
de l'apolipoprotéine E (E4 variant), considérée comme
l'un des moins controversés des facteurs putatifs de
susceptibilité des SCA en dépit de certaines inconstances de
certaines
cohortes,106
était significativement associée (P=0.04) parmi les cas ayant
une hyperlipidémie (4.1%) vs témoins sans hyperlipidémie
(1.6%).
Les résultats faux-positifs des études antérieures
sont une explication possible de la différence entre nos observations
et celles des autres. Cette question a déjà été
reconnue comme étant un problème sérieux dans les
études d'association, en particulier lorsque les tailles
d'échantillon manquent de
puissance.107
Il est difficile d'identifier les vrais vs les faux positifs par analyse de la
seule
littérature.108
Une stratification non reconnue entre les cas et les témoins peut
créer de fausses
associations,109
et l'absence de témoins génomiques négatifs dans presque
toutes les études précédentes pour exclure cette
possibilité laisse cette question ouverte. Il est aussi difficile
d'évaluer l'étendue de l'effet des biais de publication et des
hypothèses multiples.
On peut discuter que les participants à notre étude
étaient différents de ceux rapportés
précédemment et que nos résultats peuvent ne pas avoir
trait à la validité des associations positives et des
sous-groupes cliniques (ex: analyses sous-stratifiées selon
l'âge, le sexe ou une variable clinique, telle que l'hypertension,
l'hyperlipidémie ou le tabagisme). Compte tenu que la grande
majorité des variants communs du génome humain remonte à
nos racines partagées en Afrique,
110 il
n'est pas improbable qu'il y ait différentes formes de variants
fonctionnels dans notre population par rapport à d'autres. Des
mutations moins fréquentes remontant à une origine ancestrale
plus récente pourraient être corrélées avec
certains variants génétiques dans une population mais pas dans
une autre. On ne sait pas jusqu'à quel point les profils de
déséquilibre de l'association peuvent expliquer nos
observations, mais la population de notre étude est tout à fait
typique d'un contexte mélangé européen prévalent
aux Etats-Unis.
Une autre possibilité est que l'effet des variants du risque soit
différent dans des contextes génétiques
différents; si ceci est vrai, l'absence de généralisation
des résultats limitera sérieusement leur application en
clinique. Le fait que nous n'avons pu répliquer des associations
positives chez les participants à l'étude dans des séries
consécutives largement représentatives de la maladie
rencontrée en pratique clinique met une limite à
l'applicabilité possible des observations antérieures et est en
faveur de notre hypothèse qu'il est prématuré
d'extrapoler ces observations précoces aux soins cliniques
systématiques.
L'échec de l'approche du gène candidat pour identifier les
variants conférant une susceptibilité au risque de SCA incite
à envisager d'autres approches. Une approche prometteuse est de
dépister le génome entier de façon non biaisée
dans un large échantillon pour des variants qui sont significativement
associés à un risque de maladie. Couplé à la
compréhension des profils sous-jacents du déséquilibre de
liaison du génome
humain7 et la
capacité pour obtenir de façon peu onéreuse des
génotypes dans le génome, le champ évolue rapidement vers
une approche approfondie de tout le génome. Les problèmes de
cette approche incluent le nombre inconnu de variants qui jouent un rôle
sur l'effet, l'importance de l'effet transmis par chacun et l'importance
jouée par certains variants communs par rapport à des mutations
rares indépendantes dans le risque de maladie.
Indépendamment de l'approche choisie, il est clair que des cohortes
multiples et importantes, bien appariées, de cas et de témoins,
seront nécessaires pour arriver à faire des progrès
valides dans l'analyse génétique des SCA et d'autres maladies
humaines complexes. Nos observations nulles indiquent le besoin d'être
prudent dans l'interprétation des associations génétiques
des différentes populations cliniques et le besoin de validation
approfondie des facteurs génétiques de risque.
Informations sur les auteurs
Correspondance: Thomas M. Morgan, MD, Washington University School of
Medicine, McDonnell Pediatric Research Bldg, 3103, 660 Euclid Ave, St Louis,
MO 63110, (email:
morgan_t{at}kids.wustl.edu)
ou Richard P. Lifton, MD, PhD, Yale University School of Medicine, 295
Congress Ave, New Haven, CT 06510
(richard.lifton{at}yale.edu).
Contributions des auteurs: Le Dr Morgan a eu un accès complet
à toutes les données de l'étude et accepte la
responsabilité de l'intégrité des données et de
l'exactitude de l'analyse des données.
Conception et schéma de l'étude: Morgan, Lifton,
Krumholz, Spertus.
Recueil des données: Morgan, Lifton, Krumholz, Spertus.
Analyse et interprétation des données: Morgan,
Lifton, Krumholz, Spertus.
Rédaction du manuscrit: Morgan, Lifton, Krumholz,
Spertus.
Revue critique du manuscrit: Morgan, Lifton, Krumholz.
Analyse statistique: Morgan, Lifton.
Obtention du financement: Morgan, Lifton, Spertus.
Aide administrative, technique et matérielle: Lifton,
Spertus.
Supervision de l'étude: Lifton, Krumholz, Spertus.
Liens financiers: Le Dr Spertus déclare qu'il travaille pour
des comités consultatifs de l'American College of Cardiology,
l'American Heart Association, Amgen United Healthcare, Blue Cross/Blue Shield;
il a reçu des bourses du National Institutes of Health (NIH), Amgen, CV
Therapeutics, Flowcardia, et Roache Diagnostics (ayant fourni un
réagent d'un biomarqueur pour une bourse de recherche du NIH); il a par
ailleurs des intérêts dans le Seattle Angina Questionnaire, le
Kansas City Cardiomyopathy Questionnaire, le Peripheral Artery Questionnaire,
et les Health Outcomes Sciences; il a été consultant au cours
des 5 dernières années pour CV Therapeutics, Amgen, Worldheart,
et Ostuka Parmaceuticals. Le Dr Krumholz déclare qu'il a eu des
contrats de recherche avec la Colorado Foundation for Medical Care et
l'American College of Cardiology, qu'il siège dans ces comités
consultatifs pour Amgen, Alere, et United Healthcare, qu'il est un expert sur
le sujet pour VHA Inc. Les Drs Morgan et Lifton déclarent qu'ils n'ont
aucun conflit d'intérêt.
Financement/soutien: ce projet a bénéficié du
financement sous la forme de bourses de la Saint Luke's Hospital Foundation,
Kansas City, Mo, et d'une bourse R-01 HS11282-01 de l'Agency for Healthcare
Research and Quality.
Les recherches du Dr Morgan et dans le laboratoire du Dr Lifton à
Yale University ont été soutenues par le Howard Hughes Medical
Institute et par une bourse NHLBI K23 HI77272, une bourse de recherche sous
tutelle à orientation clinique du National Heart, Lung, and Blood
Institute.
Rôle du sponsor: aucune des organisations financières
n'a joué un rôle dans le schéma, la conduite de
l'étude, le recueil, la gestion, l'analyse et l'interprétation
des données, ni dans la préparation, la revue ou l'approbation
du manuscrit.
Remerciements: nous remercions Donna Buchanan, PhD, Mid-America
Heart Institute, Kansas City, Mo, pour son aide éditoriale ce qui fait
partie de son travail et pour laquelle elle n'a reçu aucune
compensation financière supplémentaire.
* Références : 13, 20, 42, 45, 72, 88, 98-100.  Affiliation des auteurs: Department of Genetics, Howard Hughes
Medical Institute, Robert Wood Johnson Clinical Scholars Program and
Department of Internal Medicine, Yale University School of Medicine, New
Haven, Conn, and Mid-America Heart Institute and University of Missouri-Kansas
City, Mo. Le Dr Morgan travaille maintenant au Department of Pediatrics,
Division of Genetics and Genomic Medicine, Washington University School of
Medicine, St Louis, Mo.
Affiliation des auteurs: Department of Genetics, Howard Hughes
Medical Institute, Robert Wood Johnson Clinical Scholars Program and
Department of Internal Medicine, Yale University School of Medicine, New
Haven, Conn, and Mid-America Heart Institute and University of Missouri-Kansas
City, Mo. Le Dr Morgan travaille maintenant au Department of Pediatrics,
Division of Genetics and Genomic Medicine, Washington University School of
Medicine, St Louis, Mo.
BIBLIOGRAPHIE
 |  |
1. Marenberg ME, Risch N, Berkman LF, Floderus B, de Faire U. Genetic
susceptibility to death from coronary heart disease in a study of twins.
N Engl J Med.1994; 330:1041
-1046.
PUBMED
2. Scheuner MT. Clinical application of genetic risk assessment
strategies for coronary artery disease: genotypes, phenotypes, and family
history. Prim Care. 2004;31
: 711-737,
xi-xii.
PUBMED
3. Casas JPCJ, Miller GJ, Hingorani AD, Humphries SE. Investigating
the genetic determinants of cardiovascular disease using candidate genes and
metaanalysis of association studies. Ann Hum Genet.2006; 70:145
-169.
PUBMED
4. Morgan TM, Coffey CS, Krumholz HM. Overestimation of genetic risks
owing to small sample sizes in cardiovascular studies. Clin
Genet. 2003;64
:7-17.
5. Yamada Y. Identification of genetic factors and development of
genetic risk diagnosis systems for cardiovascular diseases and stroke.
Circ J. 2006;70:1240
-1248.
PUBMED
6. Ioannidis JP, Ntzani EE, Trikalinos TA, Contopoulos-Ioannidis DG.
Replication validity of genetic association studies. Nat
Genet. 2001;29:306
-309.
PUBMED
7. The International HapMap Consortium. The International HapMap
Project. Nature.2003; 426:789
-796.
PUBMED
8. Lutucuta S, Ballantyne CM, Elghannam H, Gotto AM Jr, Marian AJ.
Novel polymorphisms in promoter region of ATP binding cassette transporter
gene and plasma lipids, severity, progression, and regression of coronary
atherosclerosis and response to therapy. Circ Res.2001; 88:969
-973.
FREE FULL TEXT
9. Zwarts KY, Clee SM, Zwinderman AH, et al. ABCA1 regulatory variants
influence coronary artery disease independent of effects on plasma lipid
levels. Clin Genet.2002; 61:115
-125.
PUBMED
10. Clee SM, Zwinderman AH, Engert JC, et al. Common genetic variation
in ABCA1 is associated with altered lipoprotein levels and a modified risk for
coronary artery disease. Circulation.2001; 103:1198
-1205.
FREE FULL TEXT
11. Tregouet DA, Ricard S, Nicaud V, et al. In-depth haplotype analysis
of ABCA1 gene polymorphisms in relation to plasma ApoA1 levels and myocardial
infarction. Arterioscler Thromb Vasc Biol.2004; 24:775
-781.
FREE FULL TEXT
12. Tobin MD, Braund PS, Burton PR, et al. Genotypes and haplotypes
predisposing to myocardial infarction: a multilocus case-control study.
Eur Heart J.2004; 25:459
-467.
FREE FULL TEXT
13. Zee RY, Cook NR, Reynolds R, Cheng S, Ridker PM. Haplotype analysis
of the beta2 adrenergic receptor gene and risk of myocardial infarction in
humans. Genetics.2005; 169:1583
-1587.
FREE FULL TEXT
14. Higashi K, Ishikawa T, Ito T, Yonemura A, Shige H, Nakamura H.
Association of a genetic variation in the beta 3-adrenergic receptor gene with
coronary heart disease among Japanese. Biochem Biophys Res
Commun. 1997;232:728
-730.
PUBMED
15. Sethi AA, Nordestgaard BG, Tybjaerg-Hansen A. Angiotensinogen gene
polymorphism, plasma angiotensinogen, and risk of hypertension and ischemic
heart disease: a meta-analysis. Arterioscler Thromb Vasc
Biol. 2003;23:1269
-1275.
FREE FULL TEXT
16. Fatini C, Abbate R, Pepe G, et al. Searching for a better
assessment of the individual coronary risk profile: the role of
angiotensin-converting enzyme, angiotensin II type 1 receptor and
angiotensinogen gene polymorphisms. Eur Heart J.2000; 21:633
-638.
FREE FULL TEXT
17. Helgadottir A, Manolescu A, Thorleifsson G, et al. The gene
encoding 5-lipoxygenase activating protein confers risk of myocardial
infarction and stroke. Nat Genet.2004; 36:233
-239.
PUBMED
18. Wang XL, Liu SX, McCredie RM, Wilcken DE. Polymorphisms at the
5' -end of the apolipoprotein AI gene and severity of coronary artery
disease. J Clin Invest.1996; 98:372
-377.
PUBMED
19. Reguero JR, Cubero GI, Batalla A, et al. Apolipo-protein A1 gene
polymorphisms and risk of early coronary disease.
Cardiology.1998; 90:231
-235.
PUBMED
20. Wilson PW, Schaefer EJ, Larson MG, Ordovas JM. Apolipoprotein E
alleles and risk of coronary disease: a meta-analysis. Arterioscler
Thromb Vasc Biol. 1996;16:1250
-1255.
FREE FULL TEXT
21. Lambert JC, Brousseau T, Defosse V, et al. Independent association
of an APOE gene promoter polymorphism with increased risk of myocardial
infarction and decreased APOE plasma concentrations-the ECTIM study.
Hum Mol Genet.2000; 9:57
-61.
FREE FULL TEXT
22. Aoki S, Mukae S, Itoh S, et al. The genetic factor in acute
myocardial infarction with hypertension. Jpn Circ J.2001; 65:621
-626.
PUBMED
23. Zee RY, Cook NR, Cheng S, et al. Threonine for alanine substitution
in the eotaxin (CCL11) gene and the risk of incident myocardial infarction.
Atherosclerosis.2004; 175:91
-94.
PUBMED
24. Ortlepp JR, Vesper K, Mevissen V, et al. Chemokine receptor (CCR2)
genotype is associated with myocardial infarction and heart failure in
patients under 65 years of age. J Mol Med.2003; 81:363
-367.
PUBMED
25. González P, Alvarez R, Batalla A, et al. Genetic variation
at the chemokine receptors CCR5/CCR2 in myocardial infarction.
Genes Immun.2001; 2:191
-195.
PUBMED
26. Hubacek JA, Rothe G, Pit'ha J, et al. C(-260)? T polymorphism in
the promoter of the CD14 monocyte receptor gene as a risk factor for
myocardial infarction. Circulation.1999; 99:3218
-3220.
FREE FULL TEXT
27. Kuivenhoven JA, Jukema JW, Zwinderman AH, et al; the Regression
Growth Evaluation Statin Study Group. The role of a common variant of the
cholesteryl ester transfer protein gene in the progression of coronary
atherosclerosis. N Engl J Med.1998; 338:86
-93.
PUBMED
28. Klerkx AH, Tanck MW, Kastelein JJ, et al. Haplotype analysis of the
CETP gene: not TaqIB, but the closely linked -629C? A polymorphism and a novel
promoter variant are independently associated with CETP concentration.
Hum Mol Genet.2003; 12:111
-123.
FREE FULL TEXT
29. Eriksson AL, Skrtic S, Niklason A, et al. Association between the
low activity genotype of catechol-O-methyltransferase and myocardial
infarction in a hypertensive population. Eur Heart J.2004; 25:386
-391.
FREE FULL TEXT
30. Niessner A, Marculescu R, Haschemi A, et al. Opposite effects of
CX3CR1 receptor polymorphisms V249I and T280M on the development of acute
coronary syndrome: a possible implication of fractalkine in inflammatory
activation. Thromb Haemost. 2005;93
: 949-954.
PUBMED
31. McDermott DH, Halcox JP, Schenke WH, et al. Association between
polymorphism in the chemokine receptor CX3CR1 and coronary vascular
endothelial dysfunction and atherosclerosis. Circ Res.2001; 89:401
-407.
FREE FULL TEXT
32. Patel S, Steeds R, Channer K, Samani NJ. Analysis of promoter
region polymorphism in the aldosterone synthase gene (CYP11B2) as a risk
factor for myocardial infarction. Am J Hypertens.2000; 13:134
-139.
PUBMED
33. Hautanen A, Toivanen P, Manttari M, et al. Joint effects of an
aldosterone synthase (CYP11B2) gene polymorphism and classic risk factors on
risk of myocardial infarction. Circulation.1999; 100:2213
-2218.
FREE FULL TEXT
34. Yasar U, Bennet AM, Eliasson E, et al. Allelic variants of
cytochromes P450 2C modify the risk for acute myocardial infarction.
Pharmacogenetics.2003; 13:715
-720.
PUBMED
35. Funk M, Endler G, Freitag R, et al. CYP2C9*2 and
CYP2C9*3 alleles confer a lower risk for myocardial infarction.
Clin Chem.2004; 50:2395
-2398.
FREE FULL TEXT
36. Endler G, Mannhalter C, Sunder-Plassmann H, et al. The K121Q
polymorphism in the plasma cell membrane glycoprotein 1 gene predisposes to
early myocardial infarction. J Mol Med.2002; 80:791
-795.
PUBMED
37. Schuit SC, Oei HH, Witteman JC, et al. Estrogen receptor alpha gene
polymorphisms and risk of myocardial infarction. JAMA.2004; 291:2969
-2977.
FREE FULL TEXT
38. Shearman AM, Cupples LA, Demissie S, et al. Association between
estrogen receptor alpha gene variation and cardiovascular disease.
JAMA. 2003;290:2263
-2270.
FREE FULL TEXT
39. Endler G, Mannhalter C, Sunder-Plassmann H, et al. Homozygosity for
the C? T polymorphism at nucleotide 46 in the 5' untranslated region of
the factor XII gene protects from development of acute coronary syndrome.
Br J Haematol.2001; 115:1007
-1009.
PUBMED
40. Endler G, Mannhalter C. Polymorphisms in coagulation factor genes
and their impact on arterial and venous thrombosis. Clin Chim
Acta. 2003;330:31
-55.
PUBMED
41. Rosendaal FR, Siscovick DS, Schwartz SM, Psaty BM, Raghunathan TE,
Vos HL. A common prothrombin variant (20210 G to A) increases the risk of
myocardial infarction in young women. Blood.1997; 90:1747
-1750.
FREE FULL TEXT
42. Girelli D, Russo C, Ferraresi P, et al. Polymorphisms in the factor
VII gene and the risk of myocardial infarction in patients with coronary
artery disease. N Engl J Med.2000; 343:774
-780.
PUBMED
43. Boekholdt SM, Bijsterveld NR, Moons AH, Levi M, Buller HR, Peters
RJ. Genetic variation in coagulation and fibrinolytic proteins and their
relation with acute myocardial infarction: a systematic review.
Circulation.2001; 104:3063
-3068.
FREE FULL TEXT
44. Yamada Y, Izawa H, Ichihara S, et al. Prediction of the risk of
myocardial infarction from polymorphisms in candidate genes. N Engl
J Med. 2002;347:1916
-1923.
PUBMED
45. Kenny D, Muckian C, Fitzgerald DJ, Cannon CP, Shields DC. Platelet
glycoprotein Ib alpha receptor polymorphisms and recurrent ischaemic events in
acute coronary syndrome patients. J Thromb
Thrombolysis. 2002;13:13
-19.
PUBMED
46. Douglas H, Michaelides K, Gorog DA, et al. Platelet membrane
glycoprotein Ibalpha gene -5T/C Kozak sequence polymorphism as an independent
risk factor for the occurrence of coronary thrombosis.
Heart. 2002;87:70
-74.
FREE FULL TEXT
47. Lin RC, Wang XL, Morris BJ. Association of coronary artery disease
with glucocorticoid receptor N363S variant.
Hypertension.2003; 41:404
-407.
FREE FULL TEXT
48. Hetet G, Elbaz A, Gariepy J, et al. Association studies between
haemochromatosis gene mutations and the risk of cardiovascular diseases.
Eur J Clin Invest.2001; 31:382
-388.
PUBMED
49. Yamada S, Akita H, Kanazawa K, et al. T102C polymorphism of the
serotonin (5-HT) 2A receptor gene in patients with non-fatal acute myocardial
infarction. Atherosclerosis.2000; 150:143
-148.
PUBMED
50. Jiang H, Klein RM, Niederacher D, et al. C/T polymorphism of the
intercellular adhesion molecule-1 gene (exon 6, codon 469): a risk factor for
coronary heart disease and myocardial infarction. Int J
Cardiol. 2002; 84:171
-177.
PUBMED
51. Momiyama Y, Hirano R, Taniguchi H, Nakamura H, Ohsuzu F. Effects of
interleukin-1 gene polymorphisms on the development of coronary artery disease
associated with Chlamydia pneumoniae infection. J Am Coll
Cardiol. 2001;38:712
-717.
FREE FULL TEXT
52. Georges JL, Loukaci V, Poirier O, et al; Etude CasTemoin de
l'Infarctus du Myocarde. Interleukin-6 gene polymorphisms and susceptibility
to myocardial infarction: the ECTIM study. J Mol Med.2001; 79:300
-305.
PUBMED
53. Jenny NS, Tracy RP, Ogg MS, et al. In the elderly, interleukin-6
plasma levels and the -174G>C polymorphism are associated with the
development of cardiovascular disease. Arterioscler Thromb Vasc
Biol. 2002;22:2066
-2071.
FREE FULL TEXT
54. Baroni MG, D'Andrea MP, Montali A, et al. A common mutation of the
insulin receptor substrate-1 gene is a risk factor for coronary artery
disease. Arterioscler Thromb Vasc Biol.1999; 19:2975
-2980.
FREE FULL TEXT
55. Santoso S, Kunicki TJ, Kroll H, Haberbosch W, Gardemann A.
Association of the platelet glycoprotein Ia C807T gene polymorphism with
nonfatal myocardial infarction in younger patients.
Blood. 1999;93:2449
-2453.
FREE FULL TEXT
56. Samara WM, Gurbel PA. The role of platelet receptors and adhesion
molecules in coronary artery disease. Coron Artery
Dis. 2003;14:65
-79.
PUBMED
57. Zambon A, Deeb SS, Pauletto P, Crepaldi G, Brunzell JD. Hepatic
lipase: a marker for cardiovascular disease risk and response to therapy.
Curr Opin Lipidol.2003; 14:179
-189.
PUBMED
58. Ji J, Herbison CE, Mamotte CD, Burke V, Taylor RR, van Bockxmeer
FM. Hepatic lipase gene -514 C/T polymorphism and premature coronary heart
disease. J Cardiovasc Risk.2002; 9:105
-113.
PUBMED
59. Hokanson JE. Functional variants in the lipoprotein lipase gene and
risk cardiovascular disease. Curr Opin Lipidol.1999; 10:393
-399.
PUBMED
60. Schulz S, Schagdarsurengin U, Greiser P, et al. The LDL
receptor-related protein (LRP1/A2MR) and coronary atherosclerosis-novel
genomic variants and functional consequences. Hum
Mutat. 2002;20:404
.
PUBMED
61. PROCARDIS Consortium. A trio family study showing association of
the lymphotoxin-alpha N26 (804A) allele with coronary artery disease.
Eur J Hum Genet.2004; 12:770
-774.
PUBMED
62. Ozaki K, Ohnishi Y, Iida A, et al. Functional SNPs in the
lymphotoxin-alpha gene that are associated with susceptibility to myocardial
infarction. Nat Genet. 2002;32
: 650-654.
PUBMED
63. Herrmann SM, Whatling C, Brand E, et al. Polymorphisms of the human
matrix gla protein (MGP) gene, vascular calcification, and myocardial
infarction. Arterioscler Thromb Vasc Biol.2000; 20:2386
-2393.
FREE FULL TEXT
64. Humphries SE, Martin S, Cooper J, Miller G. Interaction between
smoking and the stromelysin-1 (MMP3) gene 5A/6A promoter polymorphism
and risk of coronary heart disease in healthy men. Ann Hum
Genet. 2002;66:343
-352.
PUBMED
65. Lamblin N, Bauters C, Hermant X, Lablanche JM, Helbecque N, Amouyel
P. Polymorphisms in the promoter regions of MMP-2, MMP-3, MMP-9 and
MMP-12 genes as determinants of aneurysmal coronary artery disease.
J Am Coll Cardiol.2002; 40:43
-48.
FREE FULL TEXT
66. Klerk M, Verhoef P, Clarke R, Blom HJ, Kok FJ, Schouten EG. MTHFR
677C? T polymorphism and risk of coronary heart disease: a meta-analysis.
JAMA. 2002;288:2023
-2031.
FREE FULL TEXT
67. Ledmyr H, McMahon AD, Ehrenborg E, et al. The microsomal
triglyceride transfer protein gene-493T variant lowers cholesterol but
increases the risk of coronary heart disease.
Circulation.2004; 109:2279
-2284.
FREE FULL TEXT
68. Juo SH, Han Z, Smith JD, Colangelo L, Liu K. Common polymorphism in
promoter of microsomal triglyceride transfer protein gene influences
cholesterol, ApoB, and triglyceride levels in young African American men:
results from the coronary artery risk development in young adults (CARDIA)
study. Arterioscler Thromb Vasc Biol.2000; 20:1316
-1322.
FREE FULL TEXT
69. Hyndman ME, Bridge PJ, Warnica JW, Fick G, Parsons HG. Effect of
heterozygosity for the methionine synthase 2756 A? G mutation on the risk for
recurrent cardiovascular events. Am J Cardiol.2000; 86:1144
-1146, A1149.
PUBMED
70. Gruchala M, Ciecwierz D, Wasag B, et al. Association of the ScaI
atrial natriuretic peptide gene polymorphism with nonfatal myocardial
infarction and extent of coronary artery disease. Am Heart
J. 2003;145:125
-131.
PUBMED
71. Tatsuguchi M, Furutani M, Hinagata J, et al. Oxidized LDL receptor
gene (OLR1) is associated with the risk of myocardial infarction.
Biochem Biophys Res Commun.2003; 303:247
-250.
PUBMED
72. Gardemann A, Mages P, Katz N, Tillmanns H, Haberbosch W. The p22
phox A640G gene polymorphism but not the C242T gene variation is associated
with coronary heart disease in younger individuals.
Atherosclerosis.1999; 145:315
-323.
PUBMED
73. Inoue N, Kawashima S, Kanazawa K, Yamada S, Akita H, Yokoyama M.
Polymorphism of the NADH/NADPH oxidase p22-phox gene in patients with coronary
artery disease. Circulation.1998; 97:135
-137.
FREE FULL TEXT
74. Wenzel K, Baumann G, Felix SB. The homozygous combination of
Leu125Val and Ser563Asn polymorphisms in the PECAM1 (CD31)
gene is associated with early severe coronary heart disease. Hum
Mutat. 1999;14:545
.
PUBMED
75. Andreotti F, Porto I, Crea F, Maseri A. Inflammatory gene
polymorphisms and ischaemic heart disease: review of population association
studies. Heart.2002; 87:107
-112.
FREE FULL TEXT
76. Durrington PN, Mackness B, Mackness MI. Paraoxonase and
atherosclerosis. Arterioscler Thromb Vasc Biol.2001; 21:473
-480.
FREE FULL TEXT
77. Sanghera DK, Aston CE, Saha N, Kamboh MI. DNA polymorphisms in two
paraoxonase genes (PON1 and PON2) are associated with the
risk of coronary heart disease. Am J Hum Genet.1998; 62:36
-44.
PUBMED
78. Ridker PM, Cook NR, Cheng S, et al. Alanine for proline
substitution in the peroxisome proliferatoractivated receptor gamma-2
(PPARG2) gene and the risk of incident myocardial infarction.
Arterioscler Thromb Vasc Biol.2003; 23:859
-863.
FREE FULL TEXT
79. Cipollone F, Toniato E, Martinotti S, et al. A polymorphism in the
cyclooxygenase 2 gene as an inherited protective factor against myocardial
infarction and stroke. JAMA.2004; 291:2221
-2228.
FREE FULL TEXT
80. Ye L, Miki T, Nakura J, et al. Association of a polymorphic variant
of the Werner helicase gene with myocardial infarction in a Japanese
population. Am J Med Genet.1997; 68:494
-498.
PUBMED
81. Herrmann SM, Ricard S, Nicaud V, et al. The P-selectin gene is
highly polymorphic: reduced frequency of the Pro715 allele carriers in
patients with myocardial infarction. Hum Mol Genet.1998; 7:1277
-1284.
FREE FULL TEXT
82. Moatti D, Seknadji P, Galand C, et al. Polymorphisms of the tissue
factor pathway inhibitor (TFPI) gene in patients with acute coronary syndromes
and in healthy subjects: impact of the V264M substitution on plasma levels of
TFPI. Arterioscler Thromb Vasc Biol.1999; 19:862
-869.
FREE FULL TEXT
83. Chao TH, Li YH, Chen JH, et al. Relation of thrombomodulin gene
polymorphisms to acute myocardial infarction in patients <or =50 years of
age. Am J Cardiol.2004; 93:204
-207.
PUBMED
84. Doggen CJ, Kunz G, Rosendaal FR, et al. A mutation in the
thrombomodulin gene, 127G to A coding for Ala25Thr, and the risk of myocardial
infarction in men. Thromb Haemost.1998; 80:743
-748.
PUBMED
85. Wu KK, Aleksic N, Ahn C, Boerwinkle E, Folsom AR, Juneja H.
Thrombomodulin Ala455Val polymorphism and risk of coronary heart disease.
Circulation.2001; 103:1386
-1389.
FREE FULL TEXT
86. Topol EJ, McCarthy J, Gabriel S, et al. Single nucleotide
polymorphisms in multiple novel thrombospondin genes may be associated with
familial premature myocardial infarction. Circulation.2001; 104:2641
-2644.
FREE FULL TEXT
87. Boekholdt SM, Trip MD, Peters RJ, et al. Thrombospondin-2
polymorphism is associated with a reduced risk of premature myocardial
infarction. Arterioscler Thromb Vasc Biol.2002; 22:e24
-e27.
FREE FULL TEXT
88. Webb KE, Martin JF, Hamsten A, et al. Polymorphisms in the
thrombopoietin gene are associated with risk of myocardial infarction at a
young age. Atherosclerosis.2001; 154:703
-711.
PUBMED
89. Kolek MJ, Carlquist JF, Muhlestein JB, et al. Toll-like receptor 4
gene Asp299Gly polymorphism is associated with reductions in vascular
inflammation, angiographic coronary artery disease, and clinical diabetes.
Am Heart J.2004; 148:1034
-1040.
PUBMED
90. Padovani JC, Pazin-Filho A, Simoes MV, Marin-Neto JA, Zago MA,
Franco RF. Gene polymorphisms in the TNF locus and the risk of myocardial
infarction. Thromb Res.2000; 100:263
-269.
PUBMED
91. Poirier O, Nicaud V, Gariepy J, et al. Polymorphism R92Q of the
tumour necrosis factor receptor 1 gene is associated with myocardial
infarction and carotid intima-media thickness-the ECTIM, AXA, EVA and GENIC
Studies. Eur J Hum Genet.2004; 12:213
-219.
PUBMED
92. Alpert JS, Thygesen K, Antman E, Bassand JP. Myocardial infarction
redefined—a consensus document of the Joint European Society of
Cardiology/American College of Cardiology Committee for the redefinition of
myocardial infarction. J Am Coll Cardiol.2000; 36:959
-969.
FREE FULL TEXT
93. Braunwald E. Unstable angina: a classification.
Circulation.1989; 80:410
-414.
FREE FULL TEXT
94. Yan J, Feng J, Hosono S, Sommer SS. Assessment of multiple
displacement amplification in molecular epidemiology.
Biotechniques.2004; 37: 136-138,
140-133.
PUBMED
95. Dean FB, Hosono S, Fang L, et al. Comprehensive human genome
amplification using multiple displacement amplification. Proc Natl
Acad Sci U S A. 2002;99:5261
-5266.
FREE FULL TEXT
96. Jurinke C, van den Boom D, Cantor CR, Koster H. The use of
MassARRAY technology for high throughput genotyping. Adv Biochem
Eng Biotechnol. 2002;77:57
-74.
PUBMED
97. Jurinke C, Oeth P, van den Boom D. MALDI-TOF mass spectrometry: a
versatile tool for high-performance DNA analysis. Mol
Biotechnol. 2004;26:147
-164.
PUBMED
98. Chiodini BD, Barlera S, Franzosi MG, Beceiro VL, Introna M, Tognoni
G. APO B gene polymorphisms and coronary artery disease: a meta-analysis.
Atherosclerosis.2003; 167:355
-366.
PUBMED
99. González-Conejero R, Corral J, Roldan V, et al. A common
polymorphism in the annexin V Kozak sequence (-1C> T) increases translation
efficiency and plasma levels of annexin V, and decreases the risk of
myocardial infarction in young patients. Blood.2002; 100:2081
-2086.
FREE FULL TEXT
100. Hines LM, Stampfer MJ, Ma J, et al. Genetic variation in alcohol
dehydrogenase and the beneficial effect of moderate alcohol consumption on
myocardial infarction. N Engl J Med.2001; 344:549
-555.
PUBMED
101. Stephens M, Scheet P. Accounting for decay of linkage
disequilibrium in haplotype inference and missing-data imputation.
Am J Hum Genet.2005; 76:449
-462.
PUBMED
102. Stephens M, Smith NJ, Donnelly P. A new statistical method for
haplotype reconstruction from population data. Am J Hum
Genet. 2001;68:978
-989.
PUBMED
103. Gauderman WJ. Candidate gene association analysis for a
quantitative trait, using parentoffspring trios. Genet
Epidemiol. 2003;25:327
-338.
PUBMED
104. Gauderman WJ. Sample size requirements for matched case-control
studies of gene-environment interaction. Stat Med.2002; 21:35
-50.
PUBMED
105. Weng L, Kavaslar N, Ustaszewska A, et al. Lack of MEF2A mutations
in coronary artery disease. J Clin Invest.2005; 115:1016
-1020.
PUBMED
106. Liu S, Ma J, Ridker PM, Breslow JL, Stampfer MJ. A prospective
study of the association between APOE genotype and the risk of myocardial
infarction among apparently healthy men.
Atherosclerosis. 2003;166
: 323-329.
PUBMED
107. Freely associating. Nat Genet.1999; 22:1
-2.
PUBMED
108. Salanti G, Sanderson S, Higgins JP. Obstacles and opportunities in
meta-analysis of genetic association studies. Genet
Med. 2005;7:13
-20.
PUBMED
109. Marchini J, Cardon LR, Phillips MS, Donnelly P. The effects of
human population structure on large genetic association studies.
Nat Genet.2004; 36:512
-517.
PUBMED
110. Gabriel SB, Schaffner SF, Nguyen H, et al. The structure of
haplotype blocks in the human genome. Science.2002; 296:2225
-2229.
FREE FULL TEXT
ARTICLE EN RAPPORT
JAMA. 2007;297:1519.
Texte Complet
|